ATPase
| Adenosinatrifosfatase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Pengenal pasti | |||||||||
| Nombor EC | 3.6.1.3 | ||||||||
| Nombor CAS | 9000-83-3 | ||||||||
| Pangkalan data | |||||||||
| IntEnz | Lihat IntEnz | ||||||||
| BRENDA | Entri BRENDA | ||||||||
| ExPASy | Lihat NiceZyme | ||||||||
| KEGG | Entri KEGG | ||||||||
| MetaCyc | Laluan metabolik | ||||||||
| PRIAM | Profil | ||||||||
| Struktur PDB | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
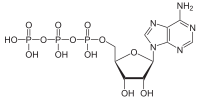
ATPase (EC 3.6.1.3, adenosina 5'-trifosfatase, adenilpirofosfatase, ATP monofosfatase, trifosfatase, SV40 T-antigen, ATP hidrolase, kompleks V (pengangkutan elektron mitokondrion), (Ca2+ + Mg2+)-ATPase, HCO3−-ATPase, adenosina trifosfatase) ialah kelas enzim yang memangkinkan penguraian ATP kepada ADP dan ion fosfat bebas[1][2][3][4][5][6] atau tindak balas songsang. Tindak balas nyahfosforilasi ini membebaskan tenaga, yang mana enzim (dalam kebanyakan kes) memanfaatkan untuk memacu tindak balas kimia lain yang tidak akan berlaku sebaliknya. Proses ini digunakan secara meluas dalam semua bentuk kehidupan yang diketahui.
Sesetengah enzim sedemikian adalah protein membran integral (terpasang dalam membran biologi), dan menggerakkan bahan terlarut merentasi membran, biasanya menentang kecerunan kepekatannya. Ini dipanggil ATPase transmembran.
Fungsi[sunting | sunting sumber]

ATPase transmembran mengimport metabolit yang diperlukan buat metabolisme sel dan mengeksport toksin, bahan buangan dan bahan terlarut yang boleh menghalang proses sel. Contoh penting ialah pam natrium-kalium (Na+/K+ ATPase) yang mengekalkan potensi membran sel. Contoh lain ialah hidrogen kalium ATPase (H+/K+ ATPase atau pam proton gastrik) yang mengasidkan kandungan perut. ATPase dipelihara secara genetik dalam haiwan; oleh itu, kardenolida yang merupakan steroid toksik yang dihasilkan oleh tumbuhan yang bertindak pada ATPase, menjadikan toksin haiwan umum dan berkesan yang bertindak bergantung kepada dos.[7]
Selain penukar, kategori lain ATPase transmembran termasuk pengangkut bersama dan pam (namun, beberapa penukar juga merupakan pam). Sebahagian enzim-enzim ini seperti Na+/K+ ATPase menyebabkan aliran bersih cas, tetapi yang lain pula tidak. Ini dipanggil pengangkut elektrogenik dan elektroneutral, masing-masing.[8]
Struktur[sunting | sunting sumber]
Motif Walker ialah motif jujukan protein dalam pengikatan nukleotida dan hidrolisis. Di luar fungsi luas ini, motif Walker boleh didapati dalam hampir semua ATPase semula jadi, dengan pengecualian ketara tirosina kinase.[9] Motif Walker biasanya membentuk motif helaian beta-pusing-heliks alfa yang disusun sendiri sebagai "sarang". Ini dianggap berlaku kerana ATPase moden berkembang daripada peptida pengikat NTP kecil yang perlu diatur sendiri.[10]
Reka bentuk protein telah dapat meniru fungsi ATPase (secara lemah) tanpa menggunakan urutan atau struktur ATPase semula jadi. Satu hal penting ialah walaupun semua ATPase semula jadi mempunyai beberapa struktur helaian beta, "ATPase alternatif" yang direka bentuk tidak mempunyai struktur helaian beta, menunjukkan bahawa fungsi penting kehidupan ini boleh dilakukan dengan jujukan dan struktur yang tidak terdapat dalam alam semula jadi.[11]
Mekanisme[sunting | sunting sumber]
ATPase (juga dipanggil F0F1-ATP sintase) ialah kompleks pemindahan cas yang memangkinkan ATP untuk melakukan sintesis ATP dengan menggerakkan ion melalui membran.[12]
Gandingan hidrolisis dan pengangkutan ATP ialah tindak balas kimia di mana bilangan molekul terlarut yang tetap diangkut untuk setiap molekul ATP yang dihidrolisiskan; bagi penukar Na+/K+, ini melibatkan tiga ion Na+ keluar dari sel, dan dua ion K+ ke dalam sel bagi setiap molekul ATP yang dihidrolisiskan.
ATPase transmembran menggunakan tenaga potensi kimia ATP dengan melakukan kerja mekanikal: ia mengangkut bahan larut dalam arah yang bertentangan dengan arah pergerakan pilihan termodinamik mereka—iaitu, dari sisi membran dengan kepekatan rendah ke sisi dengan kepekatan tinggi. Proses ini dirujuk sebagai pengangkutan aktif.
Sebagai contoh, penghalangan H+-ATPase vesikel akan mengakibatkan peningkatan pH dalam vesikel dan penurunan pH sitoplasma.
Semua ATPase berkongsi struktur asas yang sama. Setiap ATPase berputar terdiri daripada dua komponen utama: F0/A0/V0 dan F1/A1/V1. Ia disambungkan oleh 1-3 tangkai untuk mengekalkan kestabilan, mengawal putaran, dan menghalangnya daripada berputar ke arah lain. Satu tangkai digunakan untuk menghantar tork.[13] Bilangan tangkai periferi bergantung pada jenis ATPase: F-ATPase mempunyai satu, A-ATPase mempunyai dua dan V-ATPase mempunyai tiga. Domain pemangkin F1 terletak di bahagian N membran dan terlibat dalam sintesis dan degradasi ATP dan terlibat dalam fosforilasi oksidatif. Domain transmembran F0 terlibat dalam pergerakan ion merentasi membran.[12]
F0F1-ATPase bakteria terdiri daripada domain F1 larut dan domain transmembran F0 yang terdiri daripada beberapa subunit dengan stoikiometri yang berbeza-beza. Terdapat dua subunit, γ, dan ε, yang membentuk tangkai tengah dan ia dikaitkan dengan F0. F0 mengandungi oligomer subunit c dalam bentuk cincin (cincin c). Subunit α adalah berhampiran dengan subunit b2 dan membentuk tangkai yang menghubungkan subunit transmembran ke subunit α3β3 dan δ. Sintesis F-ATP adalah sama dalam rupa dan fungsi kecuali F0F1-ATP sintase mitokondrion yang mengandungi 7-9 subunit tambahan.[12]
Potensi elektrokimia inilah yang menyebabkan cincin c berputar mengikut arah jam untuk sintesis ATP. Ini menyebabkan tangkai tengah dan domain pemangkin berubah bentuk. Pemusingan cincin c menyebabkan tiga molekul ATP terhasil, dan menyebabkan H+ bergerak dari bahagian P membran ke bahagian N membran. Putaran lawan jam cincin c didorong oleh hidrolisis ATP dan pergerakan ion dari sisi N ke sisi P yang membantu mewujudkan potensi elektrokimia.[12]
Sintase ATP transmembran[sunting | sunting sumber]
ATP sintase mitokondrion dan kloroplas ialah enzim anabolik yang memanfaatkan tenaga kecerunan proton transmembran sebagai sumber tenaga untuk menambah kumpulan fosfat tak organik kepada molekul adenosina difosfat (ADP) untuk membentuk molekul adenosin trifosfat (ATP).
Enzim ini berfungsi apabila proton bergerak ke bawah kecerunan kepekatan, memberikan enzim gerakan berputar. Gerakan berputar unik ini mengikat ADP dan P bersama-sama untuk menghasilkan ATP.
Pengelasan[sunting | sunting sumber]
Terdapat pelbagai jenis ATPase, yang boleh berbeza dalam fungsi (sintesis ATP dan/atau hidrolisis), struktur (F-, V- dan A-ATPases mengandungi motor berputar) dan dalam jenis ion yang diangkut.
- ATPase berputar[14][15]
- F-ATPase (F1FO-ATPase) dalam mitokondrion, kloroplas dan membran plasma bakteria ialah pengeluar utama ATP yang menggunakan kecerunan proton yang dihasilkan oleh fosforilasi oksidatif (mitokondria) atau fotosintesis (kloroplas).[16]
- F-ATPase yang tidak mempunyai subunit delta/OSCP memindahkan ion natrium sebaliknya. Ada cadangan untuk menamakannya sebagai N-ATPase kerana ia nampaknya membentuk kumpulan berbeza yang jauh selain daripada F-ATPase biasa berbanding A-ATPase daripada V-ATPase.[17]
- V-ATPase (V1VO-ATPase) lazimnya ditemui dalam vakuol eukariot, dan memangkinkan hidrolisis ATP untuk mengangkut zat terlarut dan menurunkan pH dalam organel seperti pam proton lisosom.
- A-ATPase (A1AO-ATPase) ditemui dalam arkea dan beberapa bakteria ekstremofil. Ia terbina seperti V-ATPase, tetapi berfungsi seperti F-ATPase, terutamanya sebagai ATP sintase.
- Banyak homolog yang tidak semestinya berputar wujud.
- F-ATPase (F1FO-ATPase) dalam mitokondrion, kloroplas dan membran plasma bakteria ialah pengeluar utama ATP yang menggunakan kecerunan proton yang dihasilkan oleh fosforilasi oksidatif (mitokondria) atau fotosintesis (kloroplas).[16]
- P-ATPase (E1E2-ATPase) ditemui dalam bakteria, kulat dan dalam membran dan organel plasma eukariot, dan berfungsi untuk mengangkut pelbagai ion berbeza merentasi membran.
- E-ATPase ialah enzim permukaan sel yang menghidrolisis pelbagai NTP, termasuk ATP ekstrasel. Contohnya termasuk ekto-ATPase, CD39 dan ekto-ATP/Dase, yang kesemuanya merupakan ahli superkeluarga "GDA1 CD39".[18]
- Protein AAA ialah keluarga NTPase gelung P berbentuk cincin.
P-ATPase[sunting | sunting sumber]
P-ATPase (kadang-kadang dikenali sebagai E1-E2 ATPase) ditemui dalam bakteria serta membran dan organel plasma eukariot. Namanya sebegitu adalah kerana pemasangan singkat fosfat tak organik di sisa aspartat ketika pengaktifan. Fungsi P-ATPase adalah untuk mengangkut pelbagai sebatian berbeza seperti ion dan fosfolipid merentasi membran menggunakan hidrolisis ATP untuk tenaga. Terdapat banyak kelas P-ATPase yang berbeza yang mengangkut jenis-jenis ion tertentu. P-ATPase mungkin terdiri daripada satu atau dua polipeptida, dan biasanya boleh mengambil dua konformasi utama, E1 dan E2.
Gen manusia[sunting | sunting sumber]
- Pengangkut Na+/K+: ATP1A1, ATP1A2, ATP1A3, ATP1A4, ATP1B1, ATP1B2, ATP1B3, ATP1B4
- Pengangkut Ca++: ATP2A1, ATP2A2, ATP2A3, ATP2B1, ATP2B2, ATP2B3, ATP2B4, ATP2C1, ATP2C2
- Pengangkut Mg++: ATP3
- Penukar H+/K+: ATP4A
- Pengangkut H+, mitokondrion : ATP5A1, ATP5B, ATP5C1, ATP5C2, ATP5D, ATP5E, ATP5F1, ATP5G1, ATP5G2, ATP5G3, ATP5H, ATP5I, ATP5J2, ATP5J2, ATP5J2, ATP5J2 , ATP5J2 S , MT-ATP6, MT-ATP8
- Pengangkut H+, lisosom : ATP6AP1, ATP6AP2, ATP6V1A, ATP6V1B1, ATP6V1B2, ATP6V1C1, ATP6V1C2, ATP6V1D, ATP6V1E1, ATP6V1E2, ATP6V1E2, ATP6V1E2, ATP6V1E2, ATP6V1E2, ATP6V1E2, ATP6V1E2, ATP6V1E2 V1G3, ATP6V1H, ATP6V0A1, ATP6V0A2, ATP6V0A4, ATP6V0B, ATP6V0C, ATP6V0D1, ATP6V0D2, ATP6V0E
- Pengangkut Cu++: ATP7A, ATP7B
- Kelas I, jenis 8: ATP8A1, ATP8B1, ATP8B2, ATP8B3, ATP8B4
- Kelas II, jenis 9: ATP9A, ATP9B
- Kelas V, jenis 10: ATP10A, ATP10B, ATP10D
- Kelas VI, jenis 11: ATP11A, ATP11B, ATP11C
- Pengangkut H+/K+, bukan gastrik: ATP12A
- Jenis 13: ATP13A1, ATP13A2, ATP13A3, ATP13A4, ATP13A5
Rujukan[sunting | sunting sumber]
- ^ "Proteins controlling the helical structure of DNA". Annual Review of Biochemistry. 50: 233–60. 1981. doi:10.1146/annurev.bi.50.070181.001313. PMID 6267987.
- ^ Kielley WW (1961). "Myosin adenosine triphosphatase". Dalam Boyer PD, Lardy H, Myrbäck K (penyunting). The Enzymes. 5 (ed. 2nd). New York: Academic Press. m/s. 159–168.
- ^ "Membrane adenosine triphosphatase activities in rat pancreas". Biochimica et Biophysica Acta (BBA) - Biomembranes. 602 (2): 401–18. November 1980. doi:10.1016/0005-2736(80)90320-x. PMID 6252965.
- ^ "Proton-linked transport in chromaffin granules". Current Topics in Bioenergetics. 11: 107–147. 1981. doi:10.1016/B978-0-12-152511-8.50010-4.
- ^ "The localization of the anion-sensitive ATPase activity in corneal endothelium". Biochimica et Biophysica Acta (BBA) - Biomembranes. 644 (2): 251–6. June 1981. doi:10.1016/0005-2736(81)90382-5. PMID 6114746.
- ^ "Regulation of viral transcription and DNA replication by the SV40 large T antigen". Current Topics in Microbiology and Immunology. 93: 5–24. 1981. doi:10.1007/978-3-642-68123-3_2. ISBN 978-3-642-68125-7. PMID 6269805.
- ^ "Community-wide convergent evolution in insect adaptation to toxic cardenolides by substitutions in the Na,K-ATPase". Proceedings of the National Academy of Sciences of the United States of America. 109 (32): 13040–5. August 2012. doi:10.1073/pnas.1202111109. PMC 3420205. PMID 22826239.
- ^ "3.2: Transport in Membranes". Biology LibreTexts (dalam bahasa Inggeris). 21 January 2017. Dicapai pada 28 July 2022.
- ^ "Distantly related sequences in the alpha- and beta-subunits of ATP synthase, myosin, kinases and other ATP-requiring enzymes and a common nucleotide binding fold". EMBO J. 1 (8): 945–51. 1982. doi:10.1002/j.1460-2075.1982.tb01276.x. PMC 553140. PMID 6329717.
- ^ "Simple yet functional phosphate-loop proteins". Proceedings of the National Academy of Sciences of the United States of America. 115 (51): E11943–E11950. December 2018. doi:10.1073/pnas.1812400115. PMC 6304952. PMID 30504143. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "A Completely De Novo ATPase from Combinatorial Protein Design". Journal of the American Chemical Society. 142 (36): 15230–15234. August 2020. doi:10.1021/jacs.0c02954. PMID 32833456.
- ^ a b c d "Mechanisms of Energy Transduction by Charge Translocating Membrane Proteins". Chemical Reviews. 121 (3): 1804–1844. February 2021. doi:10.1021/acs.chemrev.0c00830. PMID 33398986.
- ^ "Structure of a Complete ATP Synthase Dimer Reveals the Molecular Basis of Inner Mitochondrial Membrane Morphology". Molecular Cell. 63 (3): 445–456. August 2016. doi:10.1016/j.molcel.2016.05.037. PMC 4980432. PMID 27373333. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Rotary ATPases--dynamic molecular machines". Current Opinion in Structural Biology. 25: 40–8. April 2014. doi:10.1016/j.sbi.2013.11.013. PMID 24878343.
- ^ "Rotary ATPases: A New Twist to an Ancient Machine". Trends in Biochemical Sciences. 41 (1): 106–116. January 2016. doi:10.1016/j.tibs.2015.10.006. PMID 26671611.
- ^ "Chemomechanical coupling mechanism of F(1)-ATPase: catalysis and torque generation". FEBS Letters. 587 (8): 1030–1035. April 2013. doi:10.1016/j.febslet.2013.01.063. PMID 23395605.
- ^ "Characterization of the N-ATPase, a distinct, laterally transferred Na+-translocating form of the bacterial F-type membrane ATPase". Bioinformatics. 26 (12): 1473–1476. June 2010. doi:10.1093/bioinformatics/btq234. PMC 2881411. PMID 20472544.
- ^ "The GDA1_CD39 superfamily: NTPDases with diverse functions". Purinergic Signalling. 7 (1): 21–45. March 2011. doi:10.1007/s11302-010-9214-7. PMC 3083126. PMID 21484095.
Pautan luar[sunting | sunting sumber]
- "ATP synthase - mesin molekul yang hebat"
- ATPase dalam Tajuk Subjek Perubatan (MeSH) di Perpustakaan Perubatan Negara AS
- Struktur mikroskop elektron ATPase daripada Bank Data EM(EMDB)
