Struktur tertier protein
Struktur tertier protein ialah bentuk tiga dimensi protein. Struktur tertier akan mempunyai rantai polipeptida tunggal "tulang belakang" dengan satu atau lebih struktur sekunder protein, yakni domain protein. Rantai sisi asid amino dan tulang belakang boleh berinteraksi dan terikat dalam beberapa cara. Interaksi dan ikatan rantai sampingan dalam protein tertentu menentukan struktur tertiernya. Struktur tertier protein ditakrifkan oleh koordinat atomnya. Koordinat ini mungkin merujuk sama ada kepada domain protein atau kepada keseluruhan struktur tertier. [1] Sebilangan struktur ini boleh mengikat antara satu sama lain, membentuk struktur kuaterner.[2]
Sejarah[sunting | sunting sumber]
Sains struktur tertier protein telah berkembang daripada satu hipotesis kepada satu definisi terperinci. Walaupun Emil Fischer telah mencadangkan protein dibuat daripada rantai polipeptida dan rantai sampingan asid amino, Dorothy Maud Wrinch merupakan saintis yang memasukkan geometri ke dalam ramalan struktur protein. Wrinch menunjukkan ini dengan model Cyclol, ramalan pertama struktur protein globul.[3] Kaedah kontemporari dapat menentukan, tanpa ramalan, struktur tertier dalam lingkungan 5 Å (0.5 nm) bagi protein kecil (<120 sisa) dan, dalam keadaan yang menggalakkan, ramalan struktur sekunder yang meyakinkan.
Penentu[sunting | sunting sumber]
Kestabilan keadaan asal[sunting | sunting sumber]
Termokestabilan[sunting | sunting sumber]
Protein yang dilipat ke dalam keadaan asalnya atau konformasi asli biasanya mempunyai tenaga bebas Gibbs yang lebih rendah (gabungan entalpi dan entropi) berbanding konformasi tak terlipat. Protein akan bercenderung ke arah konformasi tenaga rendah yang akan menentukan lipatan protein dalam persekitaran sel. Oleh kerana banyak konformasi serupa akan mempunyai tenaga yang serupa, struktur protein adalah dinamik, turun naik antara struktur serupa ini.
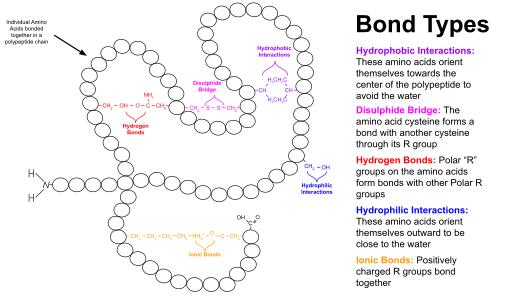
Protein globul mempunyai teras sisa asid amino hidrofobik dan kawasan permukaan sisa bercas dan hidrofilik yang terdedah air. Susunan ini boleh menstabilkan interaksi dalam struktur tertier. Sebagai contoh, dalam protein rembesan yang tidak berada dalam sitoplasma, ikatan disulfida antara sisa sisteina membantu mengekalkan struktur tertier. Ada persamaan struktur tertiari yang stabil dilihat dalam protein pelbagai fungsi dan evolusi yang pelbagai. Sebagai contoh, balang TIM, dinamakan sempena enzim triosa fosfat isomerase, ialah struktur tertier biasa seperti struktur gegelung bergelung dimer yang sangat stabil. Oleh itu, protein boleh dikelaskan mengikut struktur yang dipegangnya. Pangkalan data protein yang menggunakan klasifikasi sedemikian termasuk SCOP dan CATH.
Perangkap kinetik[sunting | sunting sumber]
Kinetik lipatan boleh memerangkap protein dalam konformasi tenaga tinggi, iaitu konformasi perantaraan tenaga tinggi menghalang akses kepada konformasi tenaga terendah. Konformasi tenaga tinggi boleh menyumbang kepada fungsi protein. Sebagai contoh, protein hemaglutinin dalam influenza ialah rantai polipeptida tunggal yang apabila diaktifkan, tetapi dipecahkan melalui proteolisis untuk membentuk dua rantai polipeptida. Kedua-dua rantai dipegang dalam konformasi tenaga tinggi. Apabila pH setempat menurun, protein mengalami penyusunan semula konformasi yang menggalakkan secara bertenaga, membolehkannya menembusi membran sel perumah.
Metakestabilan[sunting | sunting sumber]
Sesetengah struktur protein tertier mungkin wujud dalam keadaan tahan lama yang bukan merupakan keadaan paling stabil yang dijangkakan. Sebagai contoh, banyak serpin (perencat serina protease) menunjukkan metakestabilan ini. Mereka mengalami perubahan konformasi apabila gelung protein dipotong oleh protease.[4][5][6]
Protein caperon[sunting | sunting sumber]
Biasanya diandaikan bahawa keadaan asal protein juga paling stabil secara termodinamik dan protein akan mencapai keadaan asalnya, berdasarkan kinetik kimianya, sebelum ia diterjemahkan. Caperon protein dalam sitoplasma sel membantu polipeptida yang baru disintesis untuk mencapai keadaan asalnya. Sesetengah protein caperon adalah sangat spesifik dalam fungsinya seperti disulfida isomerase protein; jenis lain bersifat umum dalam fungsinya, dan boleh membantu kebanyakan protein globul, contohnya, sistem protein GroEL / GroES prokariot, dan protein kejutan haba eukariot homolog (sistem Hsp60/Hsp10).
Persekitaran sitoplasma[sunting | sunting sumber]
Ramalan struktur tertiari protein bergantung terhadap pengetahuan struktur primer protein, dan perbandingan kemungkinan struktur tertier ramalan dengan struktur tertiari yang diketahui dalam bank data protein. Ini hanya mengambil kira persekitaran sitoplasma yang hadir ketika sintesis protein sehingga takat persekitaran sitoplasma yang serupa mungkin juga telah mempengaruhi struktur protein yang direkodkan dalam bank data.
Pengikatan ligan[sunting | sunting sumber]
Struktur protein seperti enzim mungkin berubah apabila ligan aslinya diikat seperti kofaktor. Dalam kes ini, struktur protein yang terikat pada ligan dikenali sebagai struktur holo, manakala protein yang tidak terikat mempunyai struktur apo.[7]
Struktur yang distabilkan dengan pembentukan ikatan lemah antara rantai sisi asid amino ditentukan oleh lipatan rantai polipeptida dalam dirinya sendiri (sisa tak berkutub terletak di dalam protein, manakala sisa polar terletak terutamanya di luar). Penyampulan protein membawa protein lebih dekat. Pemerolehan struktur tertiari membawa kepada pembentukan poket dan tapak yang sesuai untuk pengecaman dan pengikatan molekul tertentu (biopengkhususan).
Penentuan[sunting | sunting sumber]
Pengetahuan tentang struktur tertier protein globul larut adalah lebih maju daripada protein membran kerana yang pertama lebih mudah dipelajari dengan teknologi yang ada.
Kristalografi sinar-X[sunting | sunting sumber]
Penghabluran sinar-X ialah alat yang paling biasa digunakan untuk menentukan struktur protein. Ia memberikan resolusi tinggi bagi struktur tetapi ia tidak memberikan maklumat tentang fleksibiliti konformasi protein.
NMR[sunting | sunting sumber]
NMR protein memberikan resolusi struktur protein yang agak rendah. Ia terhad kepada protein yang lebih kecil. Walau bagaimanapun, ia boleh memberikan maklumat tentang perubahan konformasi protein dalam larutan.
Mikroskopi elektron kriogen[sunting | sunting sumber]
Mikroskopi elektron kriogen (krio-EM) boleh memberi maklumat tentang kedua-dua struktur tertier dan kuaternari protein. Ia amat sesuai bagi protein besar dan kompleks simetri subunit protein.
Interferometri pengkutuban duaan[sunting | sunting sumber]
Interferometri pengkutuban duaan memberikan maklumat pelengkap tentang protein yang ditangkap permukaan. Ia membantu dalam menentukan perubahan struktur dan konformasi dari semasa ke semasa.
Projek[sunting | sunting sumber]
Algoritma ramalan[sunting | sunting sumber]
Projek Folding@home di Universiti Stanford ialah usaha penyelidikan pengkomputeran teragih yang menggunakan kira-kira 5 petaFLOPS (≈10 x86 petaFLOPS) pengkomputeran yang tersedia. Ia bertujuan untuk mencari algoritma yang secara konsisten, dan meramalkan struktur tertier dan kuaternari protein berdasarkan urutan asid amino protein dan keadaan sel.[8][9][10]
Penyakit pengumpulan protein[sunting | sunting sumber]
Penyakit pengumpulan protein seperti penyakit Alzheimer dan penyakit Huntington, dan penyakit prion seperti ensefalopati spongiforma lembu (lembu gila) boleh difahami dengan lebih baik dengan membina (dan membina semula) model penyakit. Ini dilakukan dengan mencetuskan penyakit terhadap haiwan makmal, seperti dengan memberikan toksin seperti MPTP untuk menyebabkan penyakit Parkinson, atau melalui manipulasi genetik.[11][12] Ramalan struktur protein ialah cara baharu untuk mencipta model penyakit yang mungkin dapat menyingkirkan keperluan penggunaan haiwan.[13]
Projek Pengambilan Struktur Tertier Protein (CoMOGrad)[sunting | sunting sumber]
Corak pemadanan dalam struktur tertier protein tertentu kepada sejumlah besar struktur tertier protein yang diketahui dan mendapatkan semula yang paling serupa dalam siri kedudukan merupakan satu bidang pusat dalam banyak penyelidikan seperti ramalan fungsi protein baru, kajian evolusi, diagnosis penyakit, penemuan ubat, reka bentuk antibodi dan lain-lain. Projek CoMOGrad di BUET ialah usaha penyelidikan untuk menggunakan kaedah yang sangat pantas dan tepat untuk mendapatkan semula struktur tertiari protein dan membangunkan alat dalam talian berdasarkan hasil penyelidikan.[14][15]
Rujukan[sunting | sunting sumber]
- ^ Branden C. and Tooze J. "Introduction to Protein Structure" Garland Publishing, New York.
- ^ Kyte, J. "Structure in Protein Chemistry." Garland Publishing, New York.
- ^ Senechal M. "I died for beauty: Dorothy Wrinch and the cultures of science." Oxford University Press, 2012.
- ^ Whisstock J (2006). "Molecular gymnastics: serpiginous structure, folding and scaffolding". Current Opinion in Structural Biology. 16 (6): 761–68. doi:10.1016/j.sbi.2006.10.005. PMID 17079131.
- ^ Gettins PG (2002). "Serpin structure, mechanism, and function". Chem Rev. 102 (12): 4751–804. doi:10.1021/cr010170. PMID 12475206.
- ^ "Conformational changes in serpins: I. The native and cleaved conformations of alpha(1)-anti-trypsin". J Mol Biol. 296 (2): 685–99. 2000. doi:10.1006/jmbi.1999.3520. PMID 10669617.
- ^ Seeliger, D; De Groot, B. L. (2010). "Conformational transitions upon ligand binding: Holo-structure prediction from apo conformations". PLOS Computational Biology. 6 (1): e1000634. Bibcode:2010PLSCB...6E0634S. doi:10.1371/journal.pcbi.1000634. PMC 2796265. PMID 20066034.
- ^ "Folding@home." Stanford University.
- ^ "Folding@home – FAQ" Stanford University.
- ^ "Folding@home – Science." Stanford University.
- ^ Schober A (October 2004). "Classic toxin-induced animal models of Parkinson's disease: 6-OHDA and MPTP". Cell Tissue Res. 318 (1): 215–24. doi:10.1007/s00441-004-0938-y. PMID 15503155.
- ^ "Tp53 Knockout Rat". Cancer. Dicapai pada 2010-12-18.
- ^ "Feature – What is Folding and Why Does it Matter?". Diarkibkan daripada yang asal pada December 12, 2013. Dicapai pada December 18, 2010.
- ^ "Comograd :: Protein Tertiary Matching".
- ^ Karim, Rezaul; Aziz, Mohd Momin Al; Shatabda, Swakkhar; Rahman, M. Sohel; Mia, Md Abul Kashem; Zaman, Farhana; Rakin, Salman (21 August 2015). "CoMOGrad and PHOG: From Computer Vision to Fast and Accurate Protein Tertiary Structure Retrieval". Scientific Reports. 5 (1): 13275. arXiv:1409.0814. Bibcode:2015NatSR...513275K. doi:10.1038/srep13275. PMC 4543952. PMID 26293226.
Pautan luar[sunting | sunting sumber]
- Protein Data Bank
- Paparan, analisis dan letakkan struktur 3D protein
- Abjad struktur protein.
- Paparkan, analisis dan letakkan struktur 3D protein
- Kursus berasaskan WWW mengajar bioinformatik protein asas
- Penilaian Kritikal Ramalan Struktur (CASP)
- Klasifikasi Struktur Protein (SCOP)
- Klasifikasi Struktur Protein CATH
- Perisian DALI/FSSP dan pangkalan data bagi struktur protein tertindih
- Teras Struktur Invarian TOPOFIT-DB antara protein
- PDBWiki — Halaman Utama PDBWiki – laman web untuk anotasi komuniti struktur PDB.
